Biomax 35: Genome Editing mit CRISPR-Cas9
Was ist jetzt alles möglich?

© istockphoto.com/vac1; pixabay.com/dig-designer
Wer würde nicht gerne einmal einen Blick auf einen Drachen werfen wollen? In ihrem gemeinsamen Buch „How to Build a Dragon or Die Trying“ erklären der US-amerikanische Biologe und Schriftsteller Paul Knoepfler und seine 17-jährige Tochter Julie, wie sie ihren eigenen Drachen bauen würden – dank einer der größten technologischen Innovationen der jüngsten Zeit, der Genom-Editierung. Die beiden Autoren schreiben: „Während wir über die coole Wissenschaft staunten, die es schon gibt, wurde uns auch klar – inmitten unserer Drachenbau-Pläne […] –, dass die Dinge für uns katastrophal schieflaufen könnten.“ Das Buch liefert einen satirischen Blick auf die aktuell bahnbrechendste Wissenschaft. Wir sollten uns jedoch fragen, was noch Satire und was schon Realität ist?
Die Fortschritte in der genomischen Biotechnologie bieten erstmals vielleicht die Möglichkeit, lang ausgestorbene Arten – oder zumindest „Ersatz“-Arten mit Merkmalen und ökologischen Funktionen ähnlich wie die der ausgestorbenen Originale – zurückzubringen. Ein Team unter der Leitung von George Church an der Harvard University versucht, bereits ausgestorbene Mammuts wieder zum Leben zu erwecken, indem es das Erbgut seines heute noch lebenden Verwandten, des asiatischen Elefanten, Buchstabe für Buchstabe umschreibt. Das ist möglich, seit Forschern der Pennsylvania State University 2008 die erste nahezu vollständige Sequenzierung des Erbguts eines ausgestorbenen Wollhaarmammuts gelungen ist und damit theoretisch der Zugriff auf die Information für alle seine Eigenschaften. Mehr als vier Milliarden DNA-Basen wurden dafür dekodiert (s. Biomax 33) . Das Mammut eignet sich wie kaum ein anderes ausgestorbenes Wirbeltier zur Analyse seines vorzeitlichen Erbguts. Denn die Fossilien der eiszeitlichen Elefanten stammen vorwiegend aus dem Permafrostboden Sibiriens, wo sie relativ gut erhalten bleiben. Der nächste lebende Verwandte des Wollhaarmammuts ist der asiatische Elefant. Nach Erbgutanalysen von Svante Pääbo und seinem Team vom Max-Planck-Institut für evolutionäre Anthropologie haben sich der asiatische Elefant und das Wollhaarmammut vor etwa 440.000 Jahren in verschiedene Arten aufgespalten. Das Genom des Wollhaarmammuts und des asiatischen Elefanten unterscheidet sich daher „nur“ um etwa 1,4 Millionen Mutationen: ein asiatischer Elefant besteht also praktisch bereits zu 99,96 Prozent aus Wollhaarmammut.
Jurassic-Park – Von der Fiktion zur Realität?
Das Harvard Woolly Mammoth Revival-Team hat 2015 zunächst das Erbgut eines Wollhaarmammuts analysiert und dann von bestimmten Mammutgenen exakte Kopien künstlich hergestellt. Diese wurden erfolgreich in Fibroblasten-Zelllinien des asiatischen Elefanten eingebaut. „Wir haben vor allem Gene genommen, die etwas mit der Kälteresistenz zu tun haben – also Gene für langes Fell, kleinere Ohren, die Einlagerung von Unterhautfett und vor allem für Mammut-Hämoglobin“, erklärte George Church gegenüber den Medien. Es ist ein erster Erfolg, der sich aber schnell relativiert. Denn selbst wenn man sich auf das Wesentliche beschränkt: Um ein dem Mammut stark gleichendes Genom zu erhalten, müsste das Team schon ein paar hunderttausend Erbgutabschnitte ersetzen, so die vorsichtige Schätzung. Hinzu kommt: Noch kennen sie gar nicht alle Sequenzen, die für die mammuttypischen Merkmale relevant sind.
2024 verkündete das von Church mitgegründete Start-up Colossal Bioscences nun aber die weltweit erste Reprogrammierung von Elefantenzellen. Mit den induzierten pluripotenten Stammzellen (s. Biomax 10) will man zumindest den langen und komplexen Entwicklungs- und Trächtigkeitszyklus von Elefanten und damit auch Mammuts besser verstehen. Und die Forschenden können mit diesen Zellen auch Merkmale wie Wollhaarwachstum und Fettspeicherung untersuchen. Die Wiederbelebung („de-extinction“) des Mammuts bleibt einstweilen noch Zukunftsmusik.
Eine Waffe gegen Bakteriophagen
Um was für eine Technik handelt es sich eigentlich, die die Möglichkeiten der Molekularbiologie in den vergangenen Jahren so grundlegend erweitert hat und die Fantasie der Wissenschaftler beflügelt? Wir blicken zurück in das Jahr 1987: Bei der Untersuchung von E. coli-Bakterien stoßen japanische Mikrobiologen zum ersten Mal auf ungewöhnliche, sich wiederholende DNA-Sequenzen im Erbgut eines Bakteriums. „Die biologische Bedeutung dieser Sequenzen ist vollkommen unbekannt“, schreiben sie. Wenig später nimmt der spanische Mikrobiologe Francisco Mojica an der Universität von Alicante diese Sequenzen genauer unter die Lupe. Sie lassen sich vorwärts wie rückwärts lesen, wie die Palindrom-Worte „Rentner“ oder „Lagerregal“ in der menschlichen Sprache. Während diese Worte aber durchaus eine Bedeutung haben, ergeben Palindrome im Wortschatz der Genetik keinen Sinn: Sie lassen sich nicht in funktionstüchtige Proteine übersetzen. Mojica nennt diese Sequenzen Clustered Regularly Interspaced Short Palindromic Repeats – oder kurz CRISPR. 2005 entdeckt er, dass sie mit Ausschnitten aus dem Genom eines Bakteriophagen, eines für Bakterien schädlichen Virus, übereinstimmen. Erstmals äußert er die Vermutung, dass CRISPR in Bakterien die Funktion eines adaptiven Immunsystems haben könnte. Zwei Jahre später gelingt einem französischen Wissenschaftler der Firma Danisco, dem weltweit größten Hersteller von Nahrungsmittelzusätzen, bei der Untersuchung von Streptokokken, die zur Herstellung von Joghurt eingesetzt werden, tatsächlich der experimentelle Nachweis: Philippe Horvath und sein Team integrieren Ausschnitte der Phagen-DNA in den CRISPR-Abschnitt und können so tatsächlich die nächste Phagen-Attacke bekämpfen.
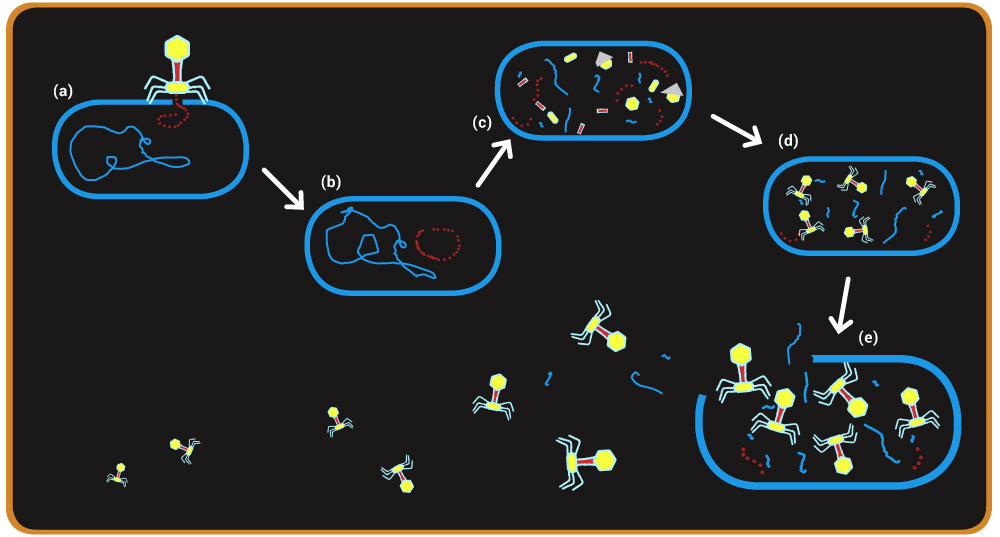
Der Bakteriophage koppelt an passende Rezeptoren an der Oberfläche des Bakteriums an (a) und injiziert die phagen-eigene DNA bzw. RNA (b). Dann beginnt die Transkription des Virusgenoms und es kommt zur Produktion der Virusbestandteile (c). Diese werden zu reifen Phagen zusammengebaut (d). Die fertigen Phagen werden durch enzymatische Auflösung der Wirtszelle befreit (e). Die Zelle platzt, und etwa 200 infektiöse Phagen werden frei.
© MPG / CC BY-NC-SA 4.0
Das letzte Puzzleteil im CRISPR-Cas-System
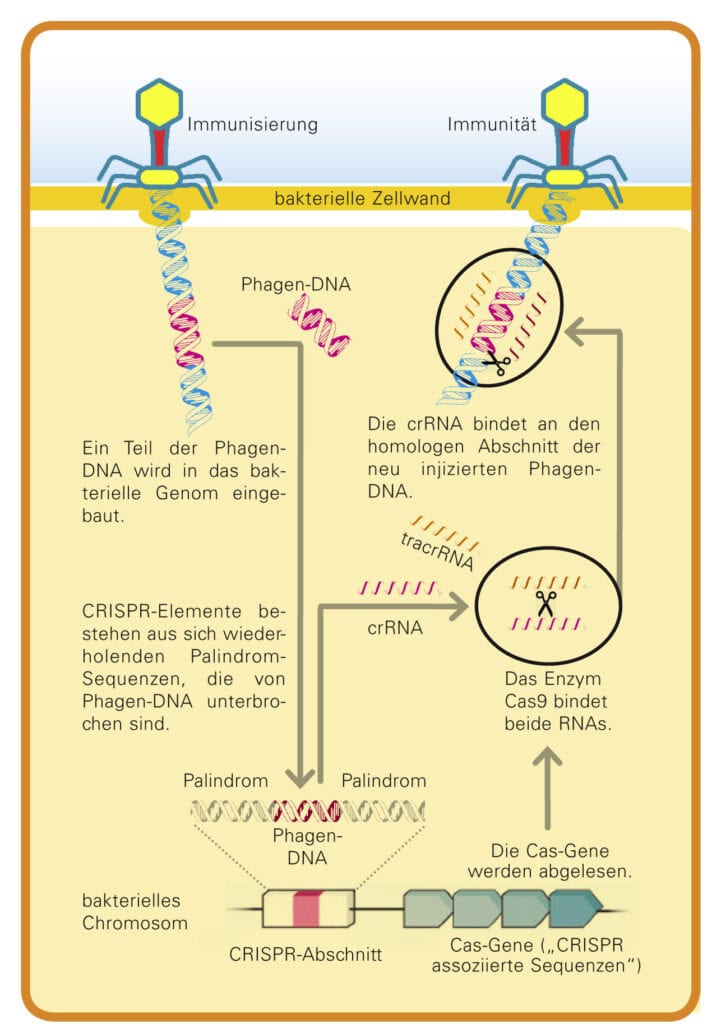
Bakterien sind ständigen Angriffen durch Bakteriophagen ausgesetzt. Denn diese sind nicht in der Lage, sich eigenständig zu vermehren. Sie müssen einen anderen Organismus kapern, in den sie ihr Erbgut einschleusen können. Die vom Phagen eingeschleusten Fremdgene programmieren das Genom des Wirtes um: Das Bakterium produziert nun keine Proteine mehr für sich selbst, sondern wird zu einer kleinen „Phagenfabrik“. Sie arbeitet so lange auf Hochtouren bis die Bakterienzelle voller Phagen ist und platzt, sodass die Phagen freigesetzt werden (Abb. A). Aber Bakterien haben Abwehrmechanismen entwickelt, um sich gegen solche Infektionen zu wehren. Wenn die Enzyme eines Bakteriums es schaffen, die injizierte Virus-DNA in kleine Stücke zu schneiden, dann kommen andere Enzyme hinzu und bauen diese Fragmente in den CRISPR-Abschnitt im bakterieneigenen Genom ein. Die seltsam aufgebauten Sequenzen stellen somit eine „Erinnerung“ an zurückliegende Virusinfektionen dar. Es ist eine Art Bibliothek sämtlicher Erreger, mit der das Bakterium schon konfrontiert worden ist. Und diese Bibliothek kann es sogar an seine Nachkommen weitergeben.
Im Jahr 2011 rätselt die französische Mikrobiologin Emmanuelle Charpentier an der Universität Umeå in Schweden darüber, wie der dahinterliegende Mechanismus der Immunabwehr funktioniert. Charpentier findet das letzte Puzzleteil im CRISPR-Cas-System, indem sie eine RNA-Sequenzierung bei einem Streptococcus-Bakterium durchführt und dabei auf zwei kurze RNAs stößt: Das Bakterium schreibt nämlich die Fremd-DNA im CRISPR-Abschnitt in ein RNA-Molekül um, CRISPR-RNA (crRNA) genannt. Diese CRISPR-RNA ist quasi ein molekularer Steckbrief, sie liefert die Erkennungssequenz, mit der das Enzym namens Cas9, eine Nuklease, die entsprechende DNA-Sequenz des eingedrungenen Virus aufspürt. Damit Cas9 aktiv werden kann, bedarf es jedoch einer zweiten kleinen RNA, die die Mikrobiologin als trans-aktivierende CRISPR-RNA (tracrRNA) bezeichnet. Erst der Komplex aus crRNA und tracrRNA führt das cas-Enzym zum Ziel: Indem Cas9 beide Stränge der Virus-DNA zerschneidet, verhindert es eine erfolgreiche Infektion durch den Bakteriophagen (Abb. B).
Zusammen mit Jennifer Doudna von der University of California in Berkeley gelingt Emmanuelle Charpentier, die heute die Max-Planck-Forschungsstelle für die Wissenschaft der Pathogene in Berlin leitet, ein Jahr später der entscheidende technologische Durchbruch: Sie fusionieren die beiden RNA-Moleküle crRNA und tracrRNA im Labor zu einem einzigen Molekül, einer sogenannten Single Guide RNA. Für den Einsatz der CRISPR/cas-Methode muss nur noch eine RNA kloniert werden. Die beiden Wissenschaftlerinnen haben damit das Funktionsprinzip von CRISPR-Cas9, in der Öffentlichkeit gerne als Gen-Schere bezeichnet, radikal vereinfacht und dafür im Jahr 2020 den Nobelpreis für Chemie erhalten.
2013 adaptiert der Biochemiker Feng Zhang, der am Broad Institute des MIT und der Harvard University forscht, CRISPR-Cas9 erfolgreich für die Genom-Editierung in eukaryotischen Zellen. Zhang und seinem Team gelingt die gezielte Genom-Editierung in kultivierten Zellen der Maus und des Menschen. Sie zeigen, dass das CRISPR-Cas-System so programmiert werden kann, dass es verschiedene genomische Abschnitte verändert. George Church, der das Wollhaarmammut wieder zum Leben erwecken will, berichtet in der gleichen Ausgabe des Fachmagazin Science über ähnliche Ergebnisse. Grundsätzlich ist Genom-Editierung nicht neu – verschiedene Techniken dafür gibt es schon seit Jahren. Was CRISPR so revolutionär macht, ist die Präzision. Und es ist unglaublich billig und einfach. Mussten Forschende früher Tausende von US-Dollar und Wochen oder Monate im Labor einsetzen, um ein Gen zu verändern, so kostet es heute noch etwa 75 US-Dollar und dauert lediglich ein paar Stunden. Und diese Technik hat bei jedem Organismus, an dem sie ausprobiert wurde, funktioniert – vom Fadenwurm über Pflanzen bis hin zum Menschen.
CRISPR ist eines der heißesten Forschungsgebiete. Im Jahr 2020 wurden über 6.000 Publikationen mit CRISPR-Bezug zur Datenbank PubMed hinzugefügt, was einem unglaublichen Durchschnitt von 16 Artikeln pro Tag entspricht. Es geht um neue Techniken zur Manipulation von Genen, Verbesserungen in der Präzision sowie weitere Arten von CRISPR-Proteinen, die ebenfalls als Gen-Editoren arbeiten. Cas13, zum Beispiel, kann RNA statt DNA editieren. „Wir sind an dem Punkt angelangt, an dem die Effizienz der Genbearbeitung auf einem Niveau liegt, das eindeutig sowohl therapeutisch als auch für eine Vielzahl anderer Anwendungen nützlich sein wird“, sagt Jennifer Doudna in einem Interview.
Und genau deshalb gibt es einen intensiven Rechtsstreit darüber, wem die Patentrechte für die CRISPR-Technologie zugesprochen werden sollen. Im Februar 2022 entschied die Berufungsinstanz des US-Patentamts, dass dem Broad Institute das Patent für CRISPR-Anwendungen bei höheren Lebewesen allein zustehe. In Europa – und ähnlich in weiteren 30 Ländern – ist die Lage jedoch anders. Das Europäische Patentamt hat die University of California in Berkeley mit Doudna und Charpentier erneut als Patentinhaberinnen bestätigt. Die Rechtslage ist verworren. Unternehmen, die an kommerziellen CRISPR-Anwendungen arbeiten, wissen nicht, an wen sie die fälligen Lizenzgebühren zahlen müssen. Die unterschiedliche Auffassung in den USA und der EU führt dazu, dass Patente häufig angefochten werden. Für akademische und andere nicht-kommerzielle Forschungsprojekte bleiben die CRISPR-Verfahren jedoch frei und ohne Lizenzgebühren nutzbar. Patentansprüche gelten hier nicht.
Genom-Editierung – Chancen und Risiken
Viele Mediziner sind überzeugt, dass sie durch das Editieren von Genen zum Beispiel Erbkrankheiten behandeln können, bei denen ein oder mehrere Gene nicht richtig funktionieren. Sie wollen es bei Mutationen anwenden, die beispielsweise die Huntington-Krankheit oder Mukoviszidose auslösen. Versuche an Mäusen haben gezeigt, dass fehlerhafte Genabschnitte, wie sie auch bei menschlichen Erbkrankheiten auftreten, durch das Editieren von Genen entfernt und die entsprechenden Krankheitsbilder behandelt werden können. Eines der größten Probleme beim Versuch, die menschliche DNA zu verändern, besteht in den sogenannten Off-Target-Effekten. Diese entstehen, wenn Cas9 ein Stück DNA schneidet, auf das es nicht programmiert wurde. Das ist wie bei der Programmierung des Navigationsgerätes im Auto beispielsweise mit der Adresse „Restaurant“. In jeder Stadt führt diese Suche zu mehreren individuellen Standorten. Aber welcher Standort ist der richtige? In der gleichen Weise wird Cas9 durch die Guide RNA zu seinem DNA-Ziel geleitet. Wenn die von der Guide RNA angegebene Adresse nicht eindeutig ist – was bei lediglich 20 Basenpaaren leicht möglich ist –, wird Cas9 an mehrere Stellen geführt, wo es die DNA schneidet. Das könnte zu unerwünschten und schwerwiegenden Nebenwirkungen, einschließlich Krebs, führen. Für jede therapeutische Anwendung beim Menschen mit Hilfe von CRISPR ist die Minimierung dieser Off-Target-Effekte daher von größter Bedeutung.
Die erste CRISPR-Therapie
Der erste Durchbruch ist nun aber gelungen: Im November 2023 erhielt eine CRISPR-Therapie in UK die weltweit erste Zulassung, im Dezember erfolgte die Zulassung in den USA und im Februar 2024 nun auch in der EU. Das Ziel der neuartigen Therapie ist die Heilung von zwei Bluterkrankungen: Beta-Thalassämie bzw. Sichelzellanämie. Bei beiden Krankheiten ist die Herstellung des Blutfarbstoffs Hämoglobin gestört, beide konnten bislang nur mit häufigen Bluttransfusionen behandelt werden, die lebensverkürzende Nebenwirkungen haben. Die Gentherapien wurden von den Biotech-Firmen Vertex Pharmaceuticals und CRISPR Therapeutics entwickelt. Emmanuelle Charpentier hat CRISPR Therapeutics gegründet und zeigt sich gegenüber der Presse glücklich, „dass CRISPR-basierte Gentherapien nach einer einzigen Behandlung einen heilsamen Effekt für Patienten mit Beta-Thalassämie und Sichelzellanämie haben.“ Die CRISPR-Therapie zielt darauf ab, die blutbildenden Zellen im Knochenmark der Patientinnen und Patienten selbst zu „ertüchtigen“: Die Zellen werden dazu gebracht, ein anderes, nämlich das fetale Hämoglobin wieder herzustellen, das sie schon vor der Geburt und in den ersten Lebenswochen produziert haben. Auch dieses Hämoglobin eignet sich gut für den Sauerstofftransport, aber es verursacht nicht die genannten Krankheitssymptome. Wie andere Gentherapien auch sind CRISPR-Therapien als Einmalbehandlung mit möglichst lang anhaltender Wirkung konzipiert. Wichtig ist es nun, genau zu beobachten, ob und gegebenenfalls welche Nebenwirkungen in den nächsten fünf bis 15 Jahren auftreten.
Exkurs: Keine Eingriffe in die menschliche Keimbahn
Der Fall des chinesischen Forschers Jiankui He, der im November 2018 berichtete, es seien Zwillinge zur Welt gekommen, bei denen er im Embryonalstadium gezielt ein Gen ausgeschaltet habe, hatte eine weltweite ethische Debatte ausgelöst. Im Oktober 2019 hat die Max-Planck-Gesellschaft in einem Positionspapier zur Genom-Editierung erklärt, dass sie aufgrund der Tragweite vererbbarer künstlicher Mutationen, wie sie bei der Genom-Editierung von Zellen der Keimbahn hervorgerufen werden, bis auf Weiteres keine Forschung zur genetischen Veränderung von Keimbahnzellen vornehmen wird. Auch der Deutsche Ethikrat hält Keimbahn-Eingriffe derzeit für zu risikoreich, will sie aber ethisch nicht grundsätzlich ausschließen.
Abbildungshinweise:
Titelbild: Elefant, Mammut © istockphoto.com/vac1; pixabay.com/dig-designer
Abb. A: Lebenszyklus Bakteriophage © Max-Planck-Gesellschaft / CC BY-NC-SA 4.0
Abb. B: Immunabwehr Bakterien © Max-Planck-Gesellschaft / CC BY-NC-SA 4.0
Abb. Exkurs: © Science Photo Library
Der Text wird unter CC BY-NC-SA 4.0 veröffentlicht.
BIOMAX Aktualisierte Ausgabe 35, Sommer 2024; Autorin: Christina Beck; Redaktion: Tanja Fendt