Biomax 33: Neandertaler mischen mit
plus Shipping Costs
Was DNA-Analysen über unsere Frühgeschichte verraten
Wer sind wir? Woher kommen wir? – das sind zentrale Fragen, die uns Menschen schon seit mehr als einem Jahrhundert beschäftigen. Spätestens seit dem Jahr 1856 als Arbeiter im Neandertal, ungefähr zwölf Kilometer östlich von Düsseldorf, in einem Steinbruch eine kleine Höhle ausräumten und dabei Reste eines Skeletts entdeckten. Über die Zuordnung der Knochenfragmente wurde lange gestritten. Die Einschätzung einiger Anatomen, dass es sich hierbei um eine Frühform des modernen Menschen handle, wurde insbesondere von dem einflussreichen deutschen Pathologen Rudolf Virchow nicht geteilt. Gegen Ende des 19. Jahrhunderts jedoch hatte sich die Auffassung, dass der Neandertaler ein Vorläufer des anatomisch modernen Menschen war, durchgesetzt.
Dank der mehr als 300 Skelettfunde ist der Neandertaler die am besten untersuchte fossile Art der Gattung Homo. Wie ähnlich die Neandertaler uns waren, ob sie einen ausgestorbenen Ast im Stammbaum der Frühmenschen darstellten und ob sich einige ihrer Gene noch heute im modernen Menschen finden, all diese Fragen ließen sich aber auf Basis rein anatomischer Untersuchungen nicht beantworten. Svante Pääbo, Direktor am Max-Planck-Institut für evolutionäre Anthropologie in Leipzig, war überzeugt, dass die Neandertalerknochen noch einen größeren Schatz bereithalten.
2005 hatte ein wissenschaftliches Konsortium unter Beteiligung von Pääbos Arbeitsgruppe das Genom des Schimpansen sequenziert und nachgewiesen, dass sich nur etwas mehr als ein Prozent der Nucleotide in den DNA-Sequenzen, die der moderne Mensch mit dem Schimpansen gemeinsam hat, unterscheiden. „Die Neandertaler sollten uns natürlich noch viel näher stehen“, sagt Pääbo. „Wenn wir aus ihren Knochen die DNA extrahieren und dann analysieren könnten“, so die Überlegungen des Molekularbiologen, „dann würden wir zweifellos feststellen, dass die Neandertaler-Gene den unseren sehr ähnlich sind.“ Viel spannender aber wären die Unterschiede: „Unter den winzigen Abweichungen, die wir erwarteten, sollten auch genau jene sein, die uns von allen unseren menschlichen Vorläufern unterscheiden und die die biologische Basis dafür gewesen sind, dass der moderne Mensch eine vollkommen neue Entwicklungsrichtung eingeschlagen hat – kulturell und technologisch.“
Die Untersuchung alter DNA erweist sich jedoch gleich in zweierlei Hinsicht als schwierig: Der eigentliche Anteil alter DNA in einem Knochenfragment kann zwischen 100 bis weniger als 0.1 Prozent liegen. In vielen Fällen sind die Proben mit der DNA von Bakterien verunreinigt. Eine weitere Quelle für Verunreinigungen ist die DNA heutiger Menschen. Denn sie ist allgegenwärtig – wir hinterlassen unsere DNA mit kleinsten Hautschuppen etc. und kontaminieren so auch archäologische Funde. Bei der Untersuchung frühmenschlichen Erbguts ist diese Kontamination aufgrund der Ähnlichkeit der DNA-Sequenzen nur schwer zu entdecken.
Abb. A: Um Verunreinigungen der Knochen mit eigener DNA zu verhindern, müssen die Forschenden umfangreiche Vorsichtsmaßnahmen treffen.
© Frank Vinken / MPG
Um die Authentizität alter DNA-Sequenzen sicherzustellen, versuchen Forschende, Verunreinigungen an der Ausgrabungsstätte sowie bei ihren weiteren molekularbiologischen Untersuchungen zu verhindern (Abb. A) oder – wenn das nicht bzw. nicht mehr möglich ist – die Verunreinigung bei der Analyse der Sequenzdaten zu identifizieren. Dabei machen sie sich den Umstand zu Nutze, dass post mortem, also nach Eintreten des Todes, Buchstaben in der DNA ausgetauscht werden: So wird Cytosin durch Thymin ersetzt bzw. Guanin durch Adenin, wenn es sich um das Gegenstück des DNA-Stranges handelt. Außerdem steigt an beiden Enden des DNA-Moleküls der Anteil jener Cytosine, denen eine Aminogruppe abhandengekommen ist. Das Cytosin wird damit zu einem Uracil, einem Nukleotid, das normalerweise in der RNA vorkommt. Die DNA-Polymerase behandelt dieses „U“ wie ein „T“ – überproportional viele Ts in bestimmten Regionen sind daher ein ausgesprochen zuverlässiges Signal, um alte von neuer DNA zu unterscheiden.
Eine weitere Erschwernis liegt darin, dass alte DNA den chemischen Abbauprozessen schon länger ausgesetzt ist, was zu einer starken Fragmentierung führt. In den Knochenproben nimmt der Anteil von DNA-Sequenzen mit kürzeren Fragmentlängen daher zu. Mit der bestehenden Technologie zur DNA-Sequenzierung konnten diese kurzen Fragmente jedoch nicht schnell und in großer Zahl ausgelesen werden.
Ein technologischer Treiber für die Paläogenetik
Den Durchbruch brachte eine ganz neue Technologie der DNA-Sequenzierung. Das Grundprinzip des Sequenzierens ist dabei unverändert geblieben: Entlang eines abzulesenden DNA-Stückes wird eine komplementäre Sequenz hergestellt. Der Einbau erkennbarer (in den meisten Fällen mit Farbstoffen markierter) Nukleotide wird registriert und anhand der zeitlichen Abfolge der Einbauereignisse die gesuchte Sequenz ermittelt. Dieses Prinzip liegt auch der Next Generation Sequencing Technology (NGS) zugrunde – nur dass hierbei das Grundprinzip des Sequenzierens in unglaublich verdichteter, effizienter und extrem vervielfältigter Weise zur Anwendung gebracht wird. Im Rahmen von Next Generation Sequencing können so mehrere Tausend bis Millionen Sequenzierungsschritte gleichzeitig und hochgradig automatisiert ablaufen (Abb. C). Das ermöglicht einen enorm hohen Probendurchsatz, so dass ein komplettes menschliches Genom mit seinen 3,2 Milliarden Buchstaben, für das das Human Genome Project noch 10 Jahre und hunderte Labore weltweit brauchte, inzwischen innerhalb weniger Tage von einem einzigen Labor sequenziert werden kann!
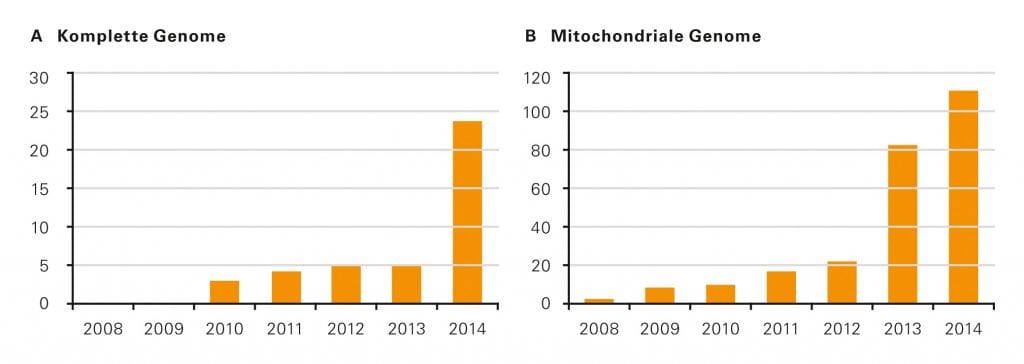
Abb. B: Gesamtzahl der publizierten vollständigen frühgeschichtlichen Humangenome.
© Investig Genet 6, 4 (2015). https://doi.org/10.1186/s13323-015-0020-4
Mittels NGS kann nun auch sehr alte, stark fragmentierte DNA mit Fragmenten, die kürzer als 60 oder 70 Basenpaare sind, sehr effektiv sequenziert werden. In Folge dessen setzte förmlich ein Boom bei der Sequenzierung alter DNA ein (Abb. B). Anfang 2006 präsentierten Stephan Schuster von der Pennsylvania State University und seine kanadischen Kollegen das 13 Millionen Basenpaare umfassende Kerngenom eines ausgestorbenen Wollmammuts. „Wir waren ein wenig enttäuscht, dass wir nicht als Erste die Sequenz einer alten DNA mit der neuen Sequenzierungstechnik aufgeklärt hatten“, berichtet Pääbo. Schließlich besaß seine Arbeitsgruppe schon seit Monaten die Daten aus den von ihr untersuchten Mammut- und Höhlenbärknochen. „Wir hatten aber weitere Analysen und Experimente durchgeführt, um ein möglichst vollständiges Bild zu veröffentlichen – die anderen hingegen wollten einfach nur schneller sein.“ Die Leipziger Forscherinnen und Forscher publizierten ihre Ergebnisse im September 2006 – und begannen noch im selben Jahr mit ihrem wohl riskantesten Projekt: der Sequenzierung des Neandertaler-Genoms. „Ich wusste, dass ein Erfolg nicht so einfach zu erzielen war“, erzählt Pääbo rückblickend. „Er hing vielmehr von drei Voraussetzungen ab: von vielen Sequenzierautomaten, viel mehr Geld und geeigneten Neandertalerknochen. Nichts davon hatten wir zu Beginn.“
Vier Jahre später war das scheinbar Unmögliche wahr geworden: Pääbo und sein Team konnten im Fachmagazin Science einen ersten Entwurf der Gensequenz unseres vor rund 30.000 Jahren ausgestorbenen Verwandten präsentieren. Der Entwurf basierte auf der Analyse von mehr als einer Milliarde DNA-Fragmenten aus mehreren Neandertaler-Knochen aus Kroatien, Spanien, Russland und Deutschland. Außerdem sequenzierten die Forschenden fünf menschliche Genome europäischer, asiatischer und afrikanischer Abstammung und verglichen diese mit dem Neandertaler-Genom. Der Vergleich förderte Erstaunliches zutage: In den Genomen aller außerhalb Afrikas lebender Menschen fanden sich Spuren vom Neandertaler. „Zwischen 1,5 und 2,1 Prozent der DNA im Genom der heutigen Nicht-Afrikaner stammen vom Neandertaler“, sagt Pääbo. „Asiaten tragen sogar noch etwas mehr davon in sich.“ Das waren klare Indizien für vielfachen artfremden Sex während der Eroberung Eurasiens.
Begonnen hatte das Techtelmechtel zwischen Neandertaler und Homo sapiens vor rund 50.000 bis 80.000 Jahren, als unsere Vorfahren den afrikanischen Kontinent verließen und sich in Europa und Asien ausbreiteten, wo sie auf die Neandertaler stießen. Während dieser Zeit kam es immer wieder zur erfolgreichen Fortpflanzung zwischen den eng verwandten Arten. Fügt man alle heute noch vorhandenen Schnipsel zusammen, lassen sich 40 Prozent des einstigen Erbmaterials der Neandertaler rekonstruieren. Von dieser DNA profitierten unsere Vorfahren. Während die meisten schädlichen Neandertaler-Gene durch Selektion aussortiert wurden, setzten sich nützliche in der menschlichen Population fest. Darunter auch solche, die mit der Beschaffenheit von Haut und Haaren in Verbindung stehen. Gut möglich also, dass unsere Vorfahren ihre weiße Haut von den Neandertalern erbten. Gerade in höheren Breiten war eine helle Körperoberfläche von Vorteil, weil damit die Produktion von Vitamin D aus Sonnenlicht effizienter ist. „Indem der moderne Mensch sich mit den Ureinwohnern seiner neuen Heimat mischte, konnte er sich besser an die neue Umgebung anpassen“, vermutet Pääbo.
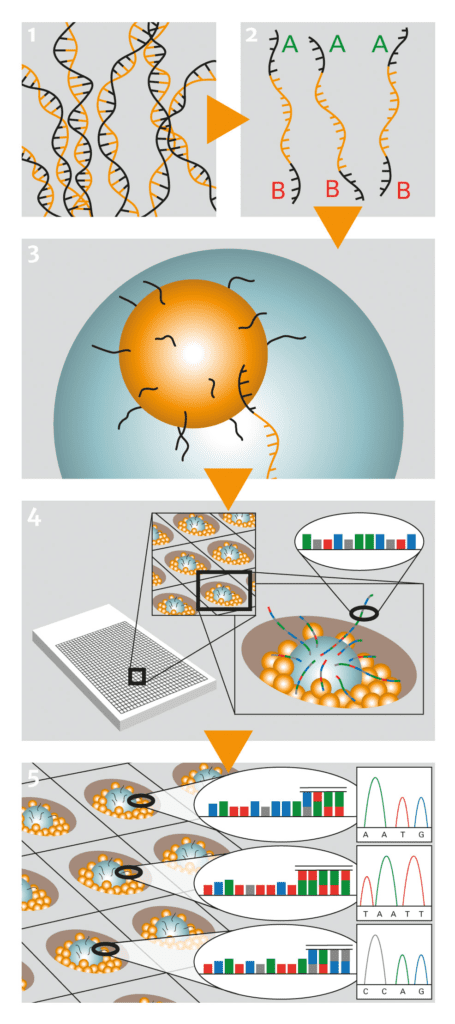
Abb. C: Next Generation Sequenzing. Svante Pääbo und sein Team sequenzierten mehr als 1 Million Basenpaare Neandertaler-DNA (1) unter Verwendung eines als Pyrosequenzierung bekannten Ansatzes. Bei diesem Verfahren wird die DNA zunächst in Einzelstränge überführt (2) und dann an mit Oligonukleotiden bestückte Mikroperlen (engl. beads) gebunden. Die DNA beladenen Mikroperlen werden zusammen mit den PCR-Reagenzien in Öl emulgiert, wobei idealerweise Emulsionströpfchen erzeugt werden, die nur eine Mikroperle enthalten (3). In dieser Umgebung werden die DNA-Stränge nun vervielfältigt (emPCR) und anschließend in die Vertiefungen einer Picotiterplatte gebracht, bei der unter jeder Pore ein Lichtleiter zu einem Detektor führt (4). Die DNA-Polymerase wird nun gewissermaßen „in Aktion“ beobachtet, wie sie nacheinander einzelne Nukleotide an den neu zu synthetisierenden DNA-Strang anhängt. Der erfolgreiche Einbau eines Nukleotids wird auf der Basis eines Fluoreszenzsignals von einem Detektor erfasst (5).
© Roche Diagnostics
Spurensuche im Genom von Homo sapiens
Und welche Auswirkungen haben die geerbten Neandertalersequenzen heute? Anhand aktueller klinischer Daten lassen sich Einflüsse auf Funktionen der Haut, des Immunsystems und des Stoffwechsels erkennen. Einige Neandertaler-Gene, die wir in uns tragen, erhöhen das Risiko, an Diabetes Typ 2 oder Morbus Crohn zu erkranken. Im Kampf gegen Krankheitserreger kann der moderne Mensch allerdings von archaischen Gensequenzen auch profitieren: Sie kodieren für drei bestimmte Immunrezeptoren und verringern damit die Neigung zu Allergien.
Während der Neandertaler in bestimmten Regionen seines Genoms noch „Schimpansen-ähnliche“ Genvarianten besitzt, tragen die meisten modernen Menschen an derselben Stelle bereits abgeleitete Genvarianten. „Genau diese Bereiche unseres Genoms könnten entscheidend zur Entwicklung des modernen Menschen beigetragen haben, weil wir hier früh in unserer evolutionären Geschichte besonders vorteilhafte Mutationen erworben haben“, sagt Pääbo. Die Veränderungen im FOXP2-Gen, das mutmaßlich die Entwicklung unsere Sprechfähigkeit orchestriert, werden hingegen von Homo sapiens und Neandertaler geteilt. Möglich also, dass der Neandertaler in dieser Hinsicht über dieselben kognitiven Fähigkeiten verfügte. Insgesamt umfasst der Katalog der genetischen Unterschiede zwischen Frühmenschen und modernen Menschen 87 Proteine und eine Handvoll microRNAs (nichtcodierende RNA-Schnipsel, die eine wichtige Rolle bei der Genregulation spielen, insbesondere beim Stummschalten von Genen).
Und dabei stehen die Wissenschaftlerinnen und Wissenschaftler erst am Anfang, die funktionellen Folgen bestimmter genetischer Änderungen zu verstehen. So haben die Max-Planck-Teams zusammen mit Kollegen von den Universitäten Barcelona und Leipzig nicht nur die DNA-Sequenz einer frühmenschlichen Genvariante analysiert, sondern auch das entsprechende Protein hergestellt und seine Eigenschaften untersucht. Dabei fanden sie heraus, dass die Aktivität einer bestimmten Genvariante des Melanocortin-Rezeptors bei zwei Neandertalern deutlich reduziert war. Genvarianten mit einer ähnlich verringerten Aktivität sind auch beim modernen Menschen bekannt – mit sichtbaren Folgen: Ihr Träger hat eine rote Haarfarbe. Die Forschenden gehen daher davon aus, dass auch ein Teil der Neandertaler möglicherweise rote Haare besaß. Die Befunde der neuen Paläogenetik führen zu einer ganz neuen Sicht auf die evolutionären Prozesse, die einst den Homo sapiens hervorbrachten und ihm als letzten Vertreter seiner Gattung zum großen Auftritt auf unserem Planeten verholfen haben. Die Erkenntnisse zeigen: Die vielen Hunderttausend Jahre der Humanevolution verliefen anders, als man lange dachte. Seit es den Forschenden gelingt, die erhaltene Erbsubstanz in Knochenfunden zum Sprechen zu bringen, bröckelt die mühsam Knochen für Knochen aufgebaute Lehrmeinung. Homo sapiens steht nicht mehr als Krone der Evolution da, sondern eher als Spross diverser „Liebschaften“ in der Vorzeit.
Unsere Populationsgeschichte im Licht alter DNA
Die Vorgänger leben weiter, im heutigen Menschen, in unserem Erbgut. Im Jahre 2010 sequenzierten Svante Pääbo und sein Team DNA aus dem winzigen Fragment eines Fingerknochens, den sie in der Denisova-Höhle in Südsibirien entdeckt hatten. „Mittels Genanalysen konnten wir zeigen, dass es sich um eine bis dahin unbekannte Menschenform handelt“, erklärt Pääbo.
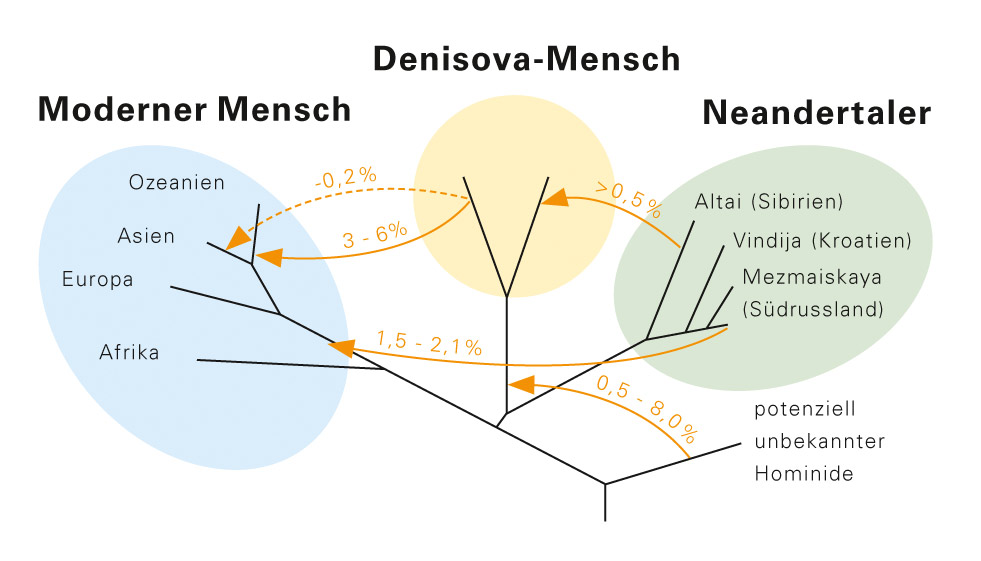
Abb. D: Die Abbildung zeigt die Richtung und den geschätzten Umfang der Genflüsse zwischen Neandertaler, Denisova-Mensch und modernem Menschen. Ob es einen direkten Genfluss vom Denisova-Menschen nach Asien gab, ist dabei ungewiss (gepunktete Linie). In drei von fünf Fällen konnten die Forscher eine Kreuzung zwischen vier verschiedenen Hominiden-Populationen nachweisen. Bei dem „potenziell unbekannten Frühmenschen“ könnte es sich um Homo erectus gehandelt haben.
© aus: K. Prüfer et al., Nature 505, 43–49, 2. Januar 2014
Und dass dieser Denisova-Mensch, wie die Forschenden ihn genannt haben, sich mit den Vorfahren der heutigen Bewohner von Australien, Neuguinea und Ostasien gepaart hat. Genomvergleiche belegen, dass es zwischen Neandertaler, Denisova und Homo sapiens zum Austausch von Genen (Genfluss) gekommen sein muss (Abb. D). „Vor diesem Hintergrund müssen wir den modernen Menschen inzwischen als Teil einer hominiden Metapopulation betrachten“, sagt Pääbo. „Einzigartig sind eigentlich nur die letzten 20.000 Jahre, in denen wir als Menschen allein auf der Welt waren.“ Und der Paläogenetiker prophezeit: „In Zukunft werden wir aus minimalen Funden sicher noch viel mehr über die Bevölkerungsgeschichte erfahren.“
Abbildungshinweise:
Titelbild: Svante Pääbo © Fank Vinken / MPG
Abb. A: Ohne Kontaminationen © Fank Vinken / MPG
Abb. B: Publizierte Humangenome © Investig Genet 6, 4 (2015). https://doi.org/10.1186/s13323-015-0020-4
Abb. C: Next Generation Sequenzing © Roche Diagnostics
Abb. D: Modell zum Genfluss © K. Prüfer et al., Nature 505, 43–49, 2. Januar 2014
Der Text wird unter CC BY-NC-SA 4.0 veröffentlicht.
BIOMAX Ausgabe 33, Winter 2016/2017, durchgesehen 07/2022; Autorin: Christina Beck; Redaktion: Tanja Fendt





