Biomax 36: Im Wettlauf mit dem Virus
plus Shipping Costs
Kein Impfstoff ohne Grundlagenforschung
© istock / kovop58
Im Dezember 2019 wird Wuhan in der chinesischen Provinz Hubei zum Zentrum eines Ausbruchs von Lungenentzündungen unbekannter Ursache. Am 7. Januar 2020 isolieren chinesische Wissenschaftler ein neuartiges Coronavirus von Patienten aus Wuhan. Nur drei Tage später, am 10. Januar publiziert die Fudan Universität in Shanghai erste Gensequenzen des neuen Erregers auf der im Internet zugänglichen weltgrößten Viren-Gendatenbank GISAID (Global Initiative on Sharing All Influenza Data); weitere Sequenzierungen von anderen Wissenschaftlern folgen. Die Gensequenz des neuartigen Coronavirus (Sars-CoV-2) ermöglicht die schnelle Entwicklung entsprechender PCR-Diagnosetests. In Deutschland vermeldet die Charité bereits am 16. Januar 2020, dass das Team um den Virologen Christian Drosten den weltweit ersten Diagnostiktest entwickelt hat. Die Geschwindigkeit der Reaktion ist rasant – und kann doch die weltweite Ausbreitung des Virus nicht verhindern.
Mittlerweile befinden wir uns im Jahr zwei der Pandemie. Ende April 2021 zählte die Johns-Hopkins-Universität in den USA über 150 Millionen Infizierte und mehr als 3 Millionen Tote weltweit. Auch nach einem Jahr der Pandemie gibt es noch kein wirksames Medikament, um eine schwere Covid-19-Erkrankung zu behandeln. Antivirale Medikamente, die bei anderen Infektionen eingesetzt werden, wirken leider nicht oder nur unzureichend. Schon kleinste Unterschiede in Aufbau und Funktionsweise der Viren beeinflussen die Wirksamkeit von Medikamenten erheblich. Die Oberflächenstrukturen von Sars-CoV-2, aber auch die Prozesse, mit denen sich das Virus in der infizierten Zelle vermehrt, werden daher im Detail erforscht, um Angriffspunkte für mögliche Therapien zu finden. Eine Struktur haben die Forschenden dabei besonders im Blick: die sogenannten Spike-Proteine an der Oberfläche des Virus, etwa 20 bis 40 an der Zahl. Das Virus braucht diese Oberflächenproteine, um Zellen infizieren zu können. Dabei bindet das Spike-Protein an einen bestimmten Rezeptor menschlicher Zellen, den ACE-Rezeptor, worauf das Virus mit der Zellmembran verschmilzt und sein Erbgut in das Zellinnere entlässt. Um sich zu vermehren, muss SARS-CoV-2 nämlich – wie alle Viren – eine Wirtszelle kapern. Nur dort findet das Virus die Maschinen und Bausteine, mit denen es sein genetisches Material vervielfachen kann, bevor es weitere Zellen infiziert (siehe BIOMAX 27).
Komplexe Moleküle in 3D
Am Max-Planck-Institut für Biophysik in Frankfurt haben Martin Beck und Gerhard Hummer mit ihren Arbeitsgruppen die Struktur des Spike-Proteins genauer untersucht. Denn aus der reinen Sequenz des Virus und der daraus abgeleiteten Abfolge der Aminosäuren lassen sich nur bedingt Erkenntnisse über mögliche „Schwachstellen“ des Virus erlangen. Um sich ein Bild zu machen, nutzt das Team die Kryo-Elektronentomografie, ein bildgebendes Verfahren zur dreidimensionalen Darstellung zellulärer Strukturen. Aufgrund seiner exponierten Lage eignet sich das Spike-Protein besonders gut als potenzielles Antigen. Die Analysen des Max-Planck-Teams zeigen, dass Antikörper an den oberen Teil des Spike-Proteins gut binden und somit das Andocken des Virus an die Zellen im Körper verhindern können. Andere Stellen des Proteins hingegen sind durch Zuckerketten vor der Erkennung durch das Immunsystem geschützt (Abb. A).
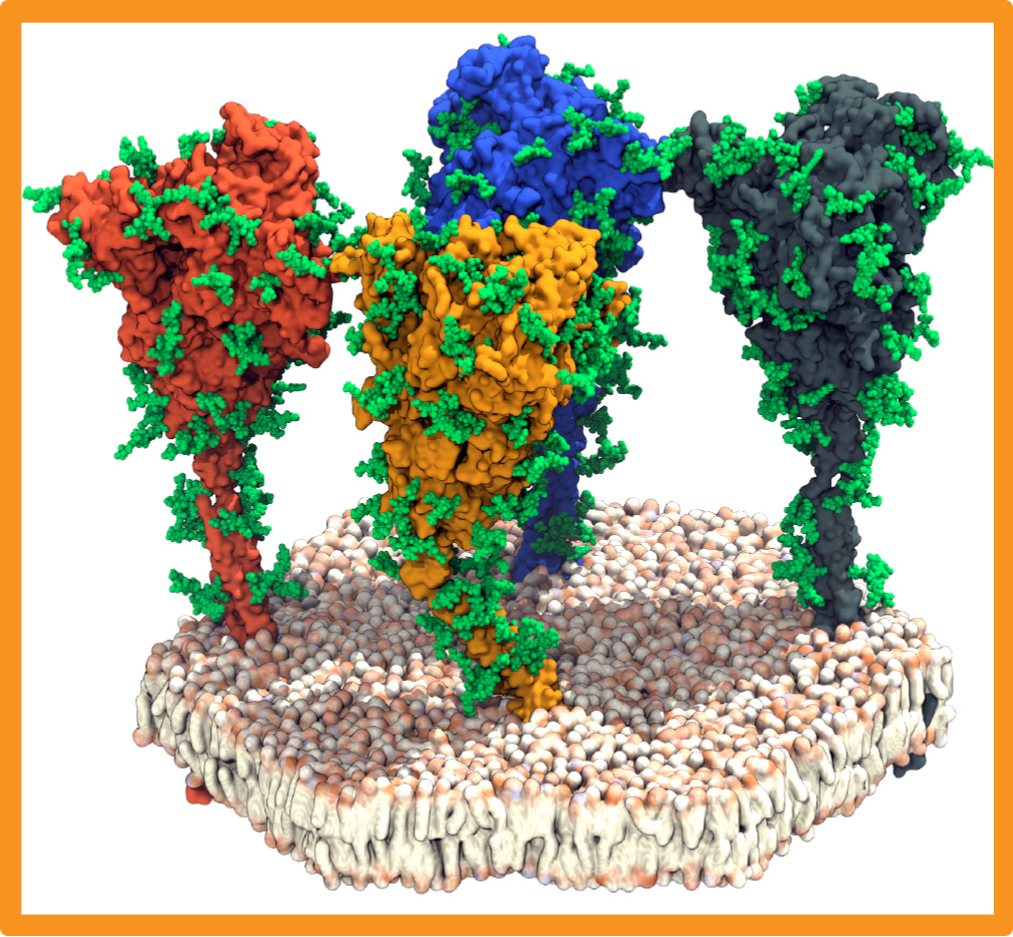
Abb. A: Andockstellen des Virus: Vier Spike-Proteine auf der Oberfläche von Sars-CoV-2 (grün: Zuckerketten). Docken diese Proteine an ACE-Rezeptoren auf der Oberfläche von Körperzellen an, kann das Virus die Zellen infizieren.
© Sikora et al., MPI für Biophysik / CC BY-NC-SA 4.0
„Bei unseren Untersuchungen haben wir festgestellt, dass der Stiel, mit dem das Spike-Protein auf der Virusoberfläche verankert ist, drei Scharniere enthält, wodurch der Proteinkopf eine unerwartete Bewegungsfreiheit erhält“, erklärt Gerhard Hummer. „Wir vermuten, dass das dem Spike-Protein ermöglicht, die Oberfläche der Wirtszelle abzutasten, um in der optimalen Position an den Rezeptor andocken zu können.“ Das Spike-Protein hat aber noch eine weitere Aufgabe: Es befördert die Fusion der Membranen des Virus und der Zelle, um das virale Genom in das Innere der Zelle zu übertragen. „Bei diesem zweiten Schritt wandelt sich die Struktur des Proteins dramatisch – von einer sogenannten Präfusions– hin zu einer Postfusionskonformation. Erst durch diese Umwandlung kann die hohe energetische Barriere der Membranfusion überwunden werden“, betont Hummer.
Auch die neuen mRNA-Impfstoffe nutzen das Spike-Protein. Anders als die sogenannten Vektor-Impfstoffe übertragen sie jedoch nicht das virale Protein selbst, sondern lediglich die Bauanleitung dafür, und machen sich damit jenes System zunutze, das unsere Zellen zur Herstellung von Proteinen verwenden (siehe BIOMAX 25) : Auf der Grundlage von Informationen in unserer DNA stellen Zellen alle lebensnotwendigen Proteine her. Um die genetische Information jedoch verarbeiten zu können, nutzen sie die sogenannte Boten- oder mRNA (engl. „messenger“) als Transkript, also als Abschrift der ursprünglichen DNA-Sequenz. mRNA-Moleküle transportieren dann die Bauanleitung für das Protein, in diesem Fall das Spike-Protein, hin zu den Ribosomen im Cytoplasma der Zelle, wo sie in die entsprechende Aminosäuresequenz übersetzt wird. Weil künstlich hergestellte mRNA selbst immunogen ist, lösen mRNA-Impfstoffe eine besonders robuste Immunantwort aus, d.h. verschiedene Arten von Immunzellen werden aktiviert und tragen zur Immunreaktion bei. Dazu gehören sowohl Antikörper-produzierende B-Zellen als auch T-Zellen, die die infizierten Zellen abtöten (Abb. B).
Die Impfstoffe sollten besonders wirksam sein, wenn die von ihnen induzierten Antikörper bei einer Infektion die Struktur des Spike-Proteins vor der Fusion der Membranen erkennen – dann können sie das Virus vor dem Eindringen in die Zelle attackieren und neutralisieren. „Deshalb haben Impfstoffentwickler wie BioNTech von Anfang an versucht, das Spike-Protein durch kleine Variationen in der mRNA in der Präfusionskonformation zu stabilisieren.“ erzählt Hummer. „Wichtige Hinweise dafür kamen aus der Strukturbiologie, die ja genau das gleiche Problem hat: eine möglichst stabile Struktur zu erzeugen, die der funktionalen Konformation entspricht.“
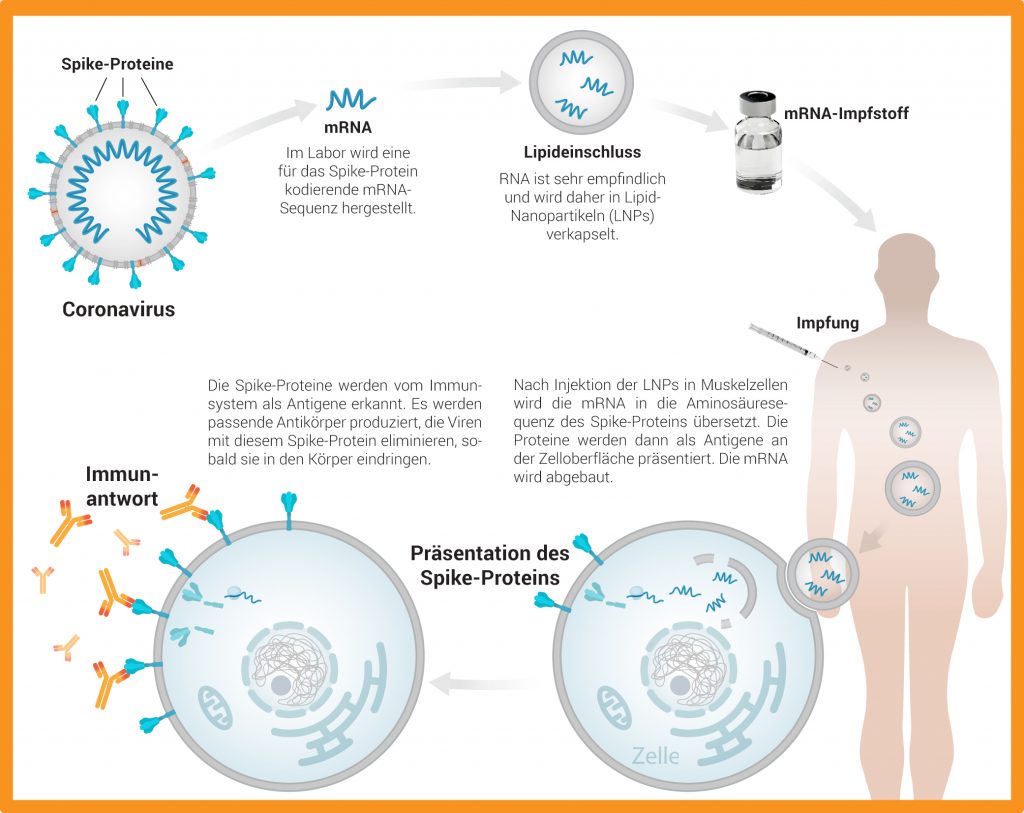
Abb. B: Wirkungsweise der mRNA-Impfstoffe
© Thomas Splettstößer / CC BY-NC-SA 4.0
Auf den Schultern von Riesen
Was heute so einfach und naheliegend erscheint, ist das Ergebnis jahrzehntelanger Grundlagenforschung, die nicht ohne Rückschläge verlief. Sie begann 1953 mit der Entdeckung der DNA-Doppelhelix. Danach zerbrachen sich die Forscher den Kopf, wie die genetische Information wohl in biologische Funktion umgesetzt wird? „Da die DNA sich im Zellkern befand und die Proteinsynthese außerhalb des Zellkerns, im Cytoplasma, stattzufinden schien, stellten wir uns vor, dass eine Kopie eines jeden aktiven Gens in das Cytoplasma geschickt werden musste. Da es hier jede Menge RNA, aber anscheinend keine DNA gab, nahmen wir an, die RNA sei dieser Bote,“ schrieb Francis Crick in seiner Autobiografie. Doch seine weiteren Schlussfolgerungen waren falsch, was u.a. daran lag, dass die Natur und Funktion von Ribosomen zu diesem Zeitpunkt noch nicht verstanden waren. Erst 1961 gelang u. a. Sydney Brenner, François Jacob und James Watson die Isolierung von mRNA und die Entwicklung eines theoretischen Konzepts. Die ersten, die die Funktion von mRNA nachweisen und im Weiteren den genetischen Code entschlüsseln konnten, waren schließlich zwei Forscher an den National Institutes of Health (NIH), Marshall Nirenberg und Heinrich Matthaei, der später Direktor am Max-Planck-Institut für experimentelle Medizin in Göttingen wurde. Die Entdeckung und Beschreibung der mRNA war somit das Produkt jahrelanger Arbeit der Science Community, die Beweise sammelten, um ein Problem zu lösen, das heute offensichtlich erscheint, aber zu der Zeit extrem schwierig war. Damit legten sie die Grundlagen für das heutige Verständnis und die weitreichenden Entwicklungen, die noch folgen sollten.
Eine körpereigene Medikamentenfabrik
mRNA könnte als Rezeptbuch für zigtausende von Proteinen dienen – daraus, so die Hoffnung, sollte sich doch ein therapeutischer Nutzen ziehen lassen: Indem man künstlich hergestellte mRNA präzise verändert und Menschen injiziert, könnte jede Zelle im Körper in eine Medikamentenfabrik auf Abruf verwandelt werden. Antigene, um gegen Infektionen zu impfen, Enzyme, um eine seltene Krankheit rückgängig zu machen, oder Wachstumsfaktoren, um beschädigtes Gewebe zu heilen. Den Grundstein für dieses Konzept legten 1990 der US-Amerikaner Jon Wolff von der Universität Wisconsin und seine Kollegen. Die Wissenschaftler injizierten nackte mRNA in Mäusemuskeln und konnten damit den Beweis für den direkten Gentransfer in vivo erbringen. Doch weitere Fortschritte ließen auf sich warten.
An der Pennsylvania State University träumte auch die ungarische Wissenschaftlerin Katalin Karikó von den scheinbar unendlichen Möglichkeiten maßgeschneiderter mRNA. Aber da Studien, die sich mit mRNA-basierten Therapeutika beschäftigten, wenig Aussicht auf Erfolg zu haben schienen, wurden sie in den Achtziger- und Neunzigerjahren kaum gefördert. Karikó musste kämpfen, um Gelder für ihre Forschung zu bekommen. Denn so einfach die Idee auch klingen mag, zwei große Herausforderungen mussten überwunden werden: erstens, die erfolgreiche Bereitstellung von mRNA. RNA ist äußerst instabil und wird von Enzymen (RNAsen) in der Umwelt und in unserem Körper leicht abgebaut. Und zweitens, die Verringerung der Immunantwort gegen die mRNA. So löst künstlich hergestellte mRNA, wenn sie in den Körper injiziert wird, eine Immunreaktion aus, infolgedessen sie zerkleinert und die Synthese der gewünschten Proteine stillgelegt wird.
Zusammen mit dem Immunologen Drew Weissman suchte Karikó nach einem Weg, um das zu verhindern. 2005 fanden sie die Lösung: Die injizierte mRNA aktiviert spezifische Rezeptoren auf den Immunzellen und löst so eine Immunantwort aus. Wird das physiologische Nukleosid Uridin durch Pseudouridin ersetzt, so bleibt die Immunantwort aufgrund einer veränderten Sekundärstruktur der mRNA aus. Das gewünschte Protein wird nun jedoch mit einer zehnmal höheren Rate produziert. Das war der Durchbruch! 2006 beantragte die Pennsylvania State University für RNA-Moleküle mit modifizierten Nukleosiden das Patent. Es ist dieses Patent, für das BioNTech und Moderna später die Lizenz erhielten. Indem sie Uridin durch zum Beispiel 2-Thio-Uridin ersetzten, konnten die Hersteller erreichen, dass zum einen nach wie vor signifikante Mengen des kodierten Proteins produziert werden, die mRNA aber gleichzeitig noch eine, wenn auch abgeschwächte Immunreaktion, insbesondere eine T-Zell-Antwort auslöst.
Auf die Verpackung kommt es an
Wie aber schleust man nun diese modifizierte RNA erfolgreich in die Zellen? mRNA-Stränge sind vergleichsweise groß und negativ geladen und können nicht einfach durch die Lipidmembranen der Zellen hindurch schlüpfen. Und wie verhindert man, dass sie durch die RNAsen in unserem Körper gleich wieder abgebaut werden? Zum Glück haben Forschende auch dafür eine Lösung gefunden. Dabei greifen sie auf eine Technologie zurück, deren Ursprünge älter sind als die Idee der mRNA-Therapie selbst: winzige Fettkügelchen, sogenannte Lipid-Nanopartikel, kurz LNPs.
LNPs nutzen einen natürlichen Prozess, die rezeptorvermittelte Endocytose, um in die Zellen zu gelangen. Man benötigt dafür positiv geladene Lipide, um die negativ geladenen Nukleinsäuren auszugleichen. Das Problem: In der Natur gibt es keine positiv geladenen Lipide. Zum einen sind sie giftig, zum anderen würden permanent positiv geladene Lipide die Zellmembranen auseinanderreißen. Die Lösung liegt in smarten Lipiden, die nur unter bestimmten Bedingungen geladen sind, sogenannte ionisierbare Lipide. Im Blut sind sie zunächst neutral. Nach der Bindung an eine Zelle wird das Nanopartikel in einer größeren Lipidblase eingekapselt, dem Endosom. Dessen Inneres hat einen sauren pH-Wert. Die Köpfe der ionisierbaren Lipide werden dadurch protoniert und sind nun positiv geladen. Die Protonierung löst eine Formveränderung des Nanopartikels aus, die vermutlich dazu führt, dass sich das LNP aus dem Endosom lösen und schließlich seine RNA-Fracht in das Cytoplasma der Zelle freisetzen kann. Einmal freigesetzt, kann die mRNA ihre Arbeit verrichten (Abb. C).
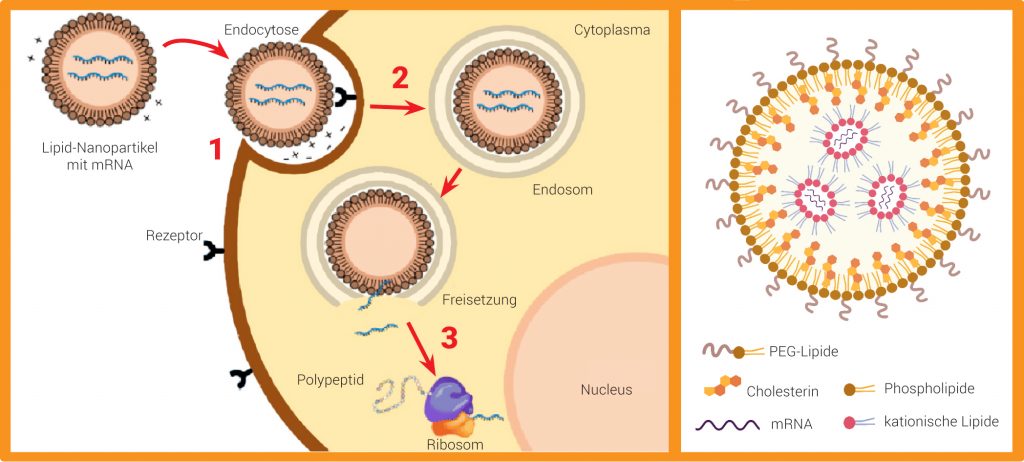
Abb. C: mRNA-Transport mithilfe von Nanopartikeln: Links: Lipid-Nanopartikel docken an spezifische Rezeptoren der Zellmembran an (1) und werden mittels Endocytose aufgenommen (2). Nach Freisetzen der mRNA aus dem Endosom startet der Translationsprozess (3). Rechts: Die LNP-Bildung erfolgt durch Selbstorganisation. PEG-Lipide schirmen sie gegen Enzyme ab; Cholesterin stabilisiert sie, indem es die Lücken zwischen den Lipiden füllt und die Aktivität der kationischen Lipide verstärkt. Phospholipide destabilisieren die Lipiddoppelschicht der Zellmembran und erhöhen so die Übertragung der LNPs in die Zelle.
© Links: verändert nach Gomes-Aguado et al; doi:10.3390/nano10020364; rechts: Max-Planck-Gesellschaft, erstellt mit BioRender.com / CC BY 4.0
Treiber für die Entwicklung dieser Lipid-Nanopartikel war zunächst eine ganz andere, 2006 mit dem Nobelpreis gekrönte Technologie: die RNA-Interferenz. Sie ermöglicht das selektive Ausschalten von Genen durch kleine RNA-Schnipsel, kurz siRNA (engl. „small interfering RNA“). Diese binden an den komplementären Abschnitt der mRNA eines Gens und schalten es damit stumm. Seit 2005 hatte die US-Biotechfirma Alnylam Pharmaceuticals, eine Ausgründung der Max-Planck-Gesellschaft zusammen mit dem Massachusetts Institute of Technology (MIT), mit verschiedenen Partnern mehr als 300 ionisierbare Lipide hergestellt, um siRNA in Zellen zu schleusen. Die Arbeit war zermürbend und Lipide, die in der Petrischale großartige Nanopartikel ergaben, versagten oft in Tierversuchen. 2010 waren sie endlich erfolgreich. Acht Jahre später brachten sie mit Onpattro nicht nur das erste auf RNA-Interferenz basierende Medikament auf den Markt, sondern gleichzeitig auch die erste zugelassene Therapie, die über Lipid-Nanopartikel verabreicht wurde.
Auch die mRNA-Impfstoffe nutzen Lipid-Nanopartikel – ohne diese Verpackung würden sie nicht funktionieren. Sie enthalten nur vier Bestandteile: ionisierbare Lipide, Polyethylenglykol (PEG)-modifizierte Lipide, sowie Phospholipide und Cholesterin. Tausende Moleküle dieser vier Komponenten kapseln die mRNA ein, schirmen sie vor zerstörerischen Enzymen ab und transportieren sie in die Zellen (Abb. C). Die verwendeten Lipide ähneln unter anderem den Phospholipiden der Zellmembran, auch Cholesterin wird zur Stabilisierung der Lipid-Doppelschicht verwendet. Die Umhüllung der mRNA mit Lipid-Nano-partikeln ist zudem der Grund dafür, warum diese Impfstoffe bei sehr niedrigen Temperaturen gelagert werden müssen: der Impfstoff von BioNTech/Pfizer bei minus 70°C, der von Moderna bei minus 20°C. Die verschiedenen Lagertemperaturen resultieren aus unterschiedlich zusammengesetzten Lipid-Umhüllungen.
Endspurt beim Impfstoff
Im Dezember 2020, noch nicht einmal ein Jahr nach der Sequenzierung des Sars-CoV-2-Genoms, erteilten die Behörden in Großbritannien und USA die erste Zulassung für den mRNA-Impfstoff von BioNTech/Pfizer. Am 21. Dezember 2020 erfolgte die Freigabe durch die Europäische Arzneimittelbehörde (EMA). Noch nie wurde ein Impfstoff so schnell entwickelt. Aber ohne jahrzehntelange Grundlagenforschung und das unermüdliche Engagement von Forscherinnen und Forschern hätten wir im Wettlauf mit dem Virus keine Chance.
Abbildungshinweise:
Titelbild: © istock / kovop58
Abb. A: Spike-Proteine © Sikora et al., MPI für Biophysik / CC BY-NC-SA 4.0
Abb. B: Wirkungsweise der mRNA-Impfstoffe © Thomas Splettstößer / CC BY-NC-SA 4.0
Abb. C: m-RNA-Transport © Links: verändert nach Gomes-Aguado et al; doi:10.3390/nano10020364 ; rechts: Max-Planck-Gesellschaft erstellt mit BioRender.com / CC BY 4.0
Der Text wird unter CC BY-NC-SA 4.0 veröffentlicht.
BIOMAX 36, Frühjahr 2021; Text: Christina Beck; Redaktion: Tanja Fendt






